Long Short-Term Memory Networks
Intro to LSTM
위 그림은 LSTM의 가장 일반적인 형태(Vanilla LSTM)다. 위 그림에서 는 입력(input)이고, 는 출력(output), 는 cell state이고 모두 벡터다. 최초 와 는 주어진다. (주로 영 벡터로 초기화하거나 임의의 벡터를 준다.) LSTM은 RNN의 특별한 케이스로 RNN처럼 LSTM 셀들을 일렬로 연결해 시퀀스(혹은 시계열) 데이터를 처리 할 수 있다. 따라서 첨자 는 번째 시퀀스를 뜻한다. 번째 셀은 입력 와 함께 이전 셀의 출력 ()과 cell state ()를 입력으로 받는다. (위 그림에서 도 LSTM의 출력이다. LSTM의 출력 로부터 원하는 크기의 벡터를 만들기 위해 프로젝션 레이어를 하나 더 둔 것이다.)
다음 그림에 3개의 LSTM cell들이 연결되어져있다.
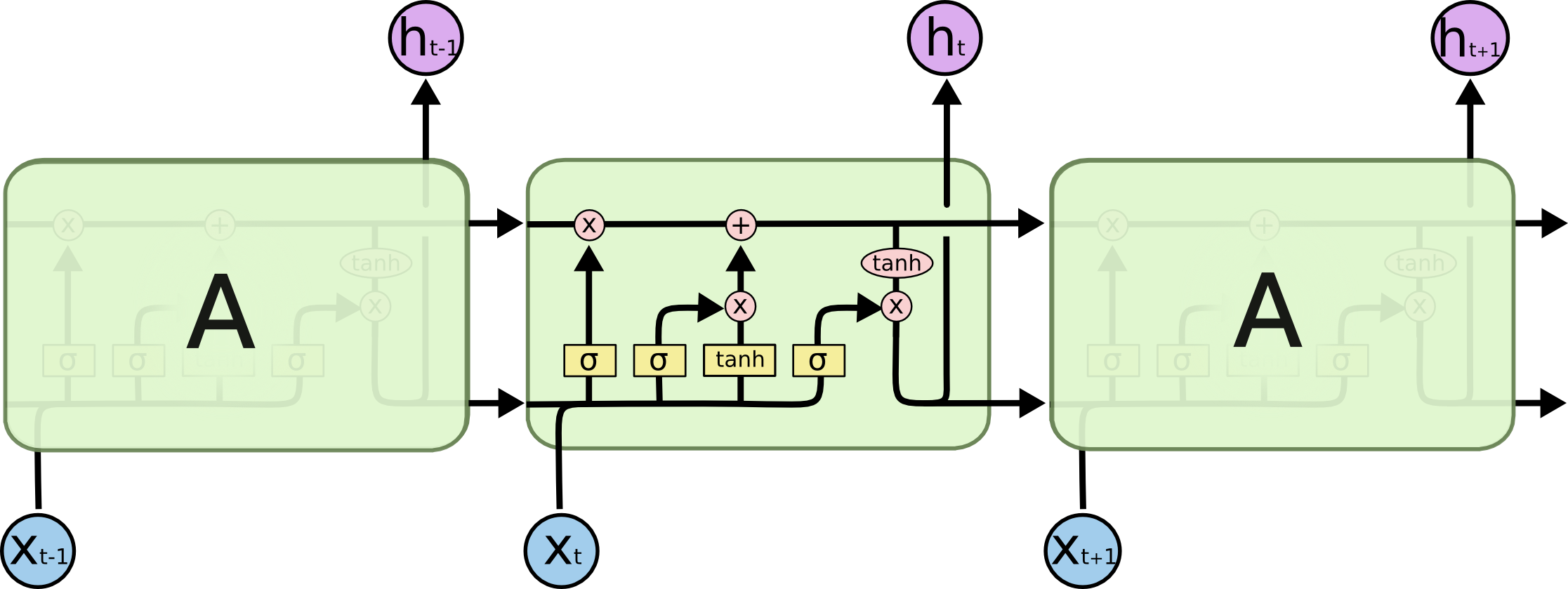
LSTM 네트워크의 핵심은 cell state인데 (위 그림에서 상단의 가로줄), 세 개의 게이트(forget gate, input gate, output gate)를 이용해서 cell state를 조절한다. 이전 셀의 cell state는 다음 셀로 입력되는데, 이때 정보를 얼마나 통과 시킬지, 또는 새로운 입력에서 어떤 정보를 취할지 등의 작업을 cell state를 통해 하게 된다.
바로 구체적으로 파고 들어가 보자.
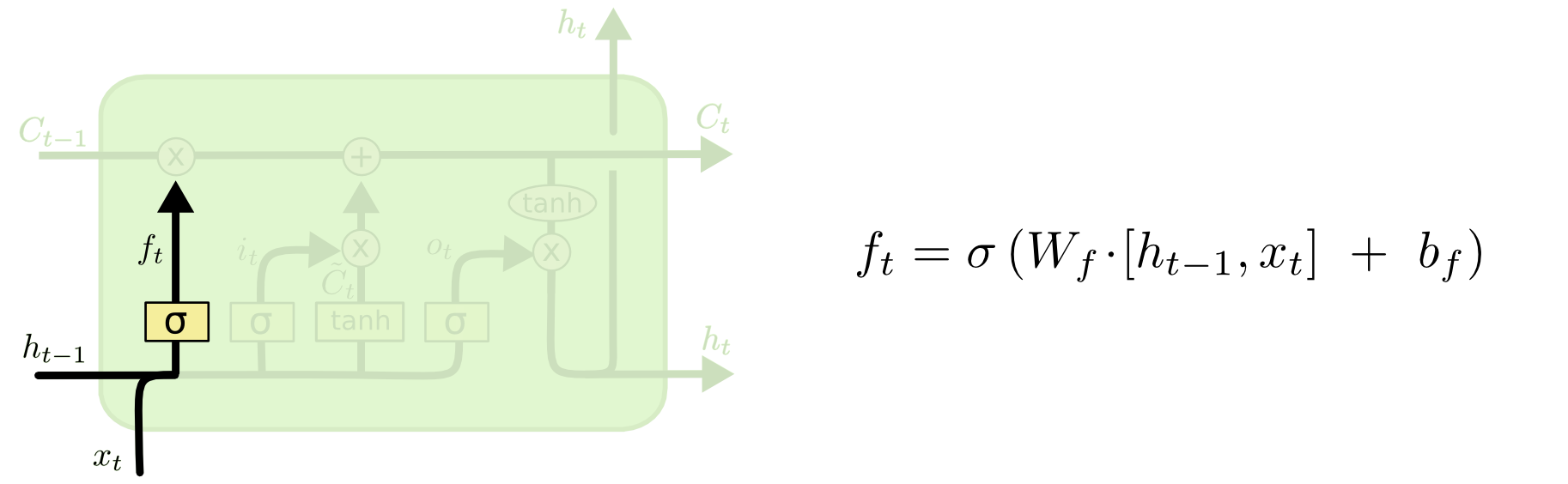 위는 forget gate이다. 과 를 입력으로 받아서 시그모이드 레이어로 출력하므로 출력이 0과 1 사이다. 0에 가까울수록 해당하는 정보가 희미해지게 되고 1에 가까우면 거의 손실없이 통과한다.
는 벡터 concatenation 이다. 즉, 이 벡터고, 가 벡터라면, 의 결과는 벡터다.
그러면 는 행렬이 되어야한다. 는 벡터다. 는 cell state 벡터의 크기(원소 수)다. 즉, cell state 벡터 는 벡터다. 시그모이드는 원소 별로(element wise) 연산을 하니까 결과적으로 는 크기의 벡터로 각 원소는 0~1 사이의 값을 갖는다.
위는 forget gate이다. 과 를 입력으로 받아서 시그모이드 레이어로 출력하므로 출력이 0과 1 사이다. 0에 가까울수록 해당하는 정보가 희미해지게 되고 1에 가까우면 거의 손실없이 통과한다.
는 벡터 concatenation 이다. 즉, 이 벡터고, 가 벡터라면, 의 결과는 벡터다.
그러면 는 행렬이 되어야한다. 는 벡터다. 는 cell state 벡터의 크기(원소 수)다. 즉, cell state 벡터 는 벡터다. 시그모이드는 원소 별로(element wise) 연산을 하니까 결과적으로 는 크기의 벡터로 각 원소는 0~1 사이의 값을 갖는다.
다음은 input gate인데 입력으로부터 어떤 정보를 얼마나 더할지 뺄지를 결정하는 역할을 한다. 이 부분은 두개의 레이어로 되어있다.
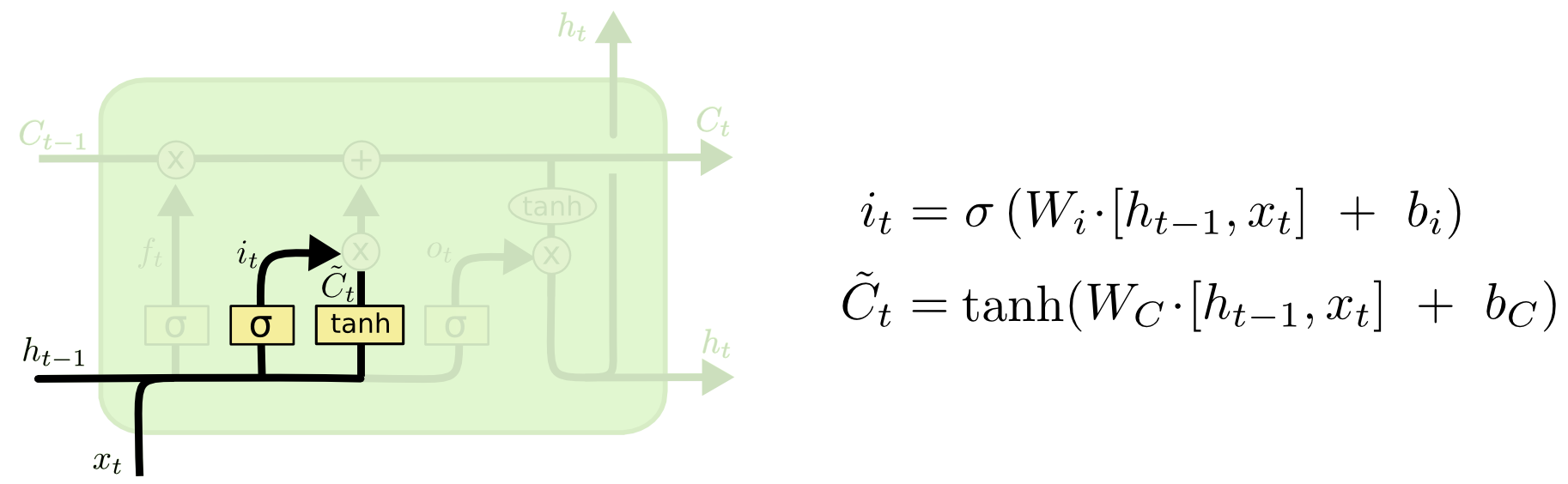 는 벡터고, 는 행렬이다. 는 벡터다. 역시 마찬가지다. 는 forget gate와 마찬가지로 시그모이드 연산을 거치므로 0과 1사이의 값을 가지며 forget gate와 비슷하게 입력으로부터 정보를 얼마나 통과 시킬지를 결정짓게 된다.
는 벡터고, 연산을 거치므로 -1에서 1사이의 값을 가진다. 이를통해 어떤 정보를 더할지 뺄지 결정하게 된다.
는 벡터고, 는 행렬이다. 는 벡터다. 역시 마찬가지다. 는 forget gate와 마찬가지로 시그모이드 연산을 거치므로 0과 1사이의 값을 가지며 forget gate와 비슷하게 입력으로부터 정보를 얼마나 통과 시킬지를 결정짓게 된다.
는 벡터고, 연산을 거치므로 -1에서 1사이의 값을 가진다. 이를통해 어떤 정보를 더할지 뺄지 결정하게 된다.
이제 새 cell state를 계산할 수 있다.
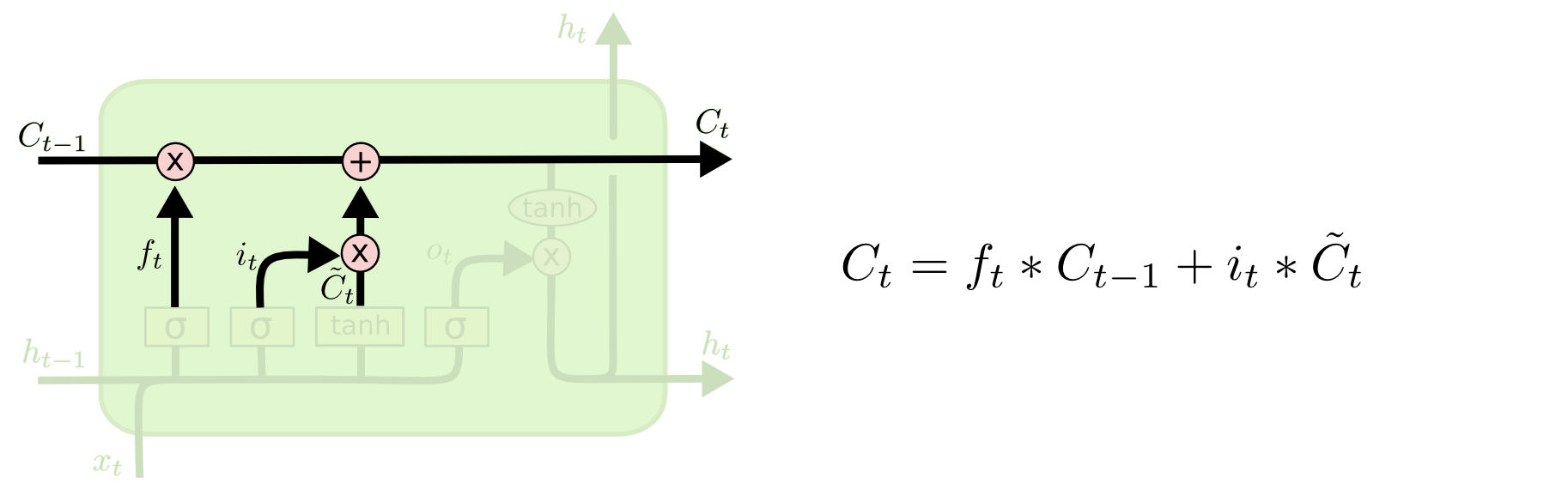 여기서 는 원소 별(element wise) 곱셈 연산이다. 이전 cell state ()에 앞서 계산한 를 곱하고, 를 더하여 새로운 cell state를 계산한다.
당연히 는 벡터다.
여기서 는 원소 별(element wise) 곱셈 연산이다. 이전 cell state ()에 앞서 계산한 를 곱하고, 를 더하여 새로운 cell state를 계산한다.
당연히 는 벡터다.
마지막으로 LSTM cell의 출력 를 계산한다. output gate이다.
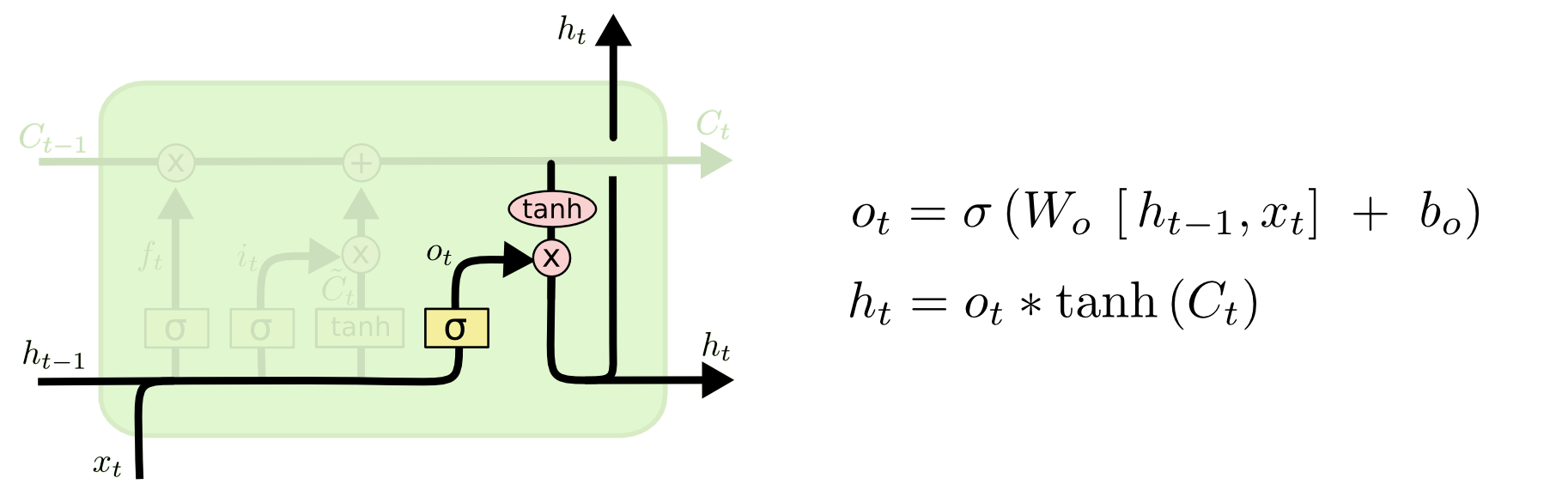 먼저 크기의 를 계산한다. 역시 는 행렬 는 벡터다. 그리고나서 를 해서 출력 를 계산한다.
먼저 크기의 를 계산한다. 역시 는 행렬 는 벡터다. 그리고나서 를 해서 출력 를 계산한다.
그런데 이때 한 가지 문제가 생긴다. 일단 라면 의 크기가 과 같기때문에 다음 LSTM 셀의 입력으로 들어 갈 수 있다. 하지만 라면 와 가 다른 크기를 가져서 문제가 생긴다. 이때는 의 크기를 로 맞춰주는 레이어를 하나 더 둠으로써 해결한다. 이 레이어를 프로젝션(projection) 레이어라고도 부른다. 즉, 로 크기를 가지는 를 계산하는 레이어를 두어 를 다음 셀의 입력으로 보낸다. (이 부분은 그림에서 빠져있다. 아마 인 경우에 대해서만 그린것 같다. 그림 출처) 는 행렬이고 는 벡터다.
LSTM 네트워크의 초기 cell state와 는 영 벡터를 쓰거나 임의의 값을 주기도 한다. 또 일련의 연결된 LSTM 셀들은 같은 웨이트를 가진다(weight sharing). 그러면 LSTM 네트워크에서 훈련시켜야할 것들은, , 그리고 프로젝션 레이어가 있다면 이다.
LSTM in Tensorflow and Numpy
여기서는 LSTM을 numpy를 이용해 구현해보고 그 계산 결과가 tensorflow의 결과와 같은지를 확인해보자. LSTM의 forward pass만 계산하여 볼 것이다. 이것의 목적은 정말 LSTM이 우리가 이해한대로 동작하는지 확인하는 것에 있다. (텐서플로에서는 모델만 만들면 알아서 backward pass (backpropagation)를 계산해서 웨이트 업데이트까지 해준다.)
4~7번 줄에서 입력 벡터의 크기는 3, cell state 벡터의 크기는 4, 출력 벡터의 크기는 5, 시퀀스 길이는 10으로 정했다.
8번줄의 forget_bias_value는 tensorflow의 LSTM에서 사용되는 변수다.
forget gate의 bias로써 디폴트로 1이 설정되어있다. 트레이닝 시작 단계에서 입력의 영향력이 줄어드는 것(forgetting)을 방지하게 해준다(참고).
10번 줄부터 31번 줄이 텐서플로로 구현한 LSTM 네트워크이다.
11~13번 줄에서 입력()과 초기 상태()를 위한 placeholder를 정의해 주었고 numpy와 precision을 맞추기위해 tf.float64를 사용하였다.
17번 줄에서 LSTM 셀을 정의하고 있다. 출력의 크기를 num_hidden으로 맞춰주기위한 num_proj=num_hidden이 보인다.
21번 줄에서 seq_len만큼 연결된 LSTM 네트워크를 dynamic_rnn 함수로 만들었고 초기 cell state()와 벡터를 넘겨주었다.
28번 줄에서 모든 시퀀스 에 대해서 인 입력 벡터와 임의의 값을 가지는 와 를 LSTM 네트워크로 넘겨주고있다.
텐서플로에서는 17번 줄처럼 initializer를 따로 정해주지 않을 경우, 랜덤으로 초기화된 weight를 가지는 LSTM 셀을 생성한다.
따라서 33~35번 줄에서 텐서플로로 생성된 LSTM의 weight 값들을 저장했다. 결과를 재현하기위해 이 값을 numpy로 구현한 LSTM에서 사용할 것이다.
40에서 77번 줄은 numpy로 구현한 LSTM 네트워크이다. 44~46번 줄에서 텐서플로 LSTM에서 사용했던 입력과 초기상태와 같은 값으로 numpy LSTM의 입력과 초기상태를 준비한다.
50번 줄에서 입력 x_np[seq]와 이전 출력 h_np를 concatenation하고 있다. 이 결과로 args의 크기(원소 수)는 num_input + num_hidden 인 벡터가 된다.
이제 텐서플로의 결과를 재현하기 위해서 텐서플로 LSTM의 weight 값과 bias 값을 가져와야한다. 51, 52번 줄에서 그 값들을 복사하고 있다. weights_tf의 0번째에는 LSTM 셀의 모든 weight들이 들어있고, 1번째에는 모든 bias들이 들어있다.
또 projection layer가 있다면 2번째에 그 weight가 들어있다 (projection layer의 경우 bias는 없다). 이때 weights_tf의 0번째에는 가 순서대로 들어있어서 크기의 행렬이 들어있다.
1번째에는 그에 상응하는 bias들이 들어있어 크기의 벡터가 들어있다. 58, 59번 줄에서 이 weight와 bias들을 이용해 행렬 연산을 하는데 이때 이용되는 트릭은 다음과 같다.
이때 이다. 61번 줄에서 이 연산의 결과를 4개로 분리하고 있다. (아래 numpy 구현에서는 행렬, 벡터 곱셉 순서가 거꾸로인 것에 주의)
63번 줄에서 np.tanh로 를 계산하고 있다.
66번 줄에서 forget_bias를 더하여 (forget gate)를 계산하고 있다.
68번 줄에서 (input gate)를 먼저 구하고 를 계산하고 있다.
69번 줄에서 (output gate)를 먼저 구하고 출력 를 계산하고 있다.
projection layer가 있을 경우 72번 줄에서 계산을 한다.
76, 77번 줄에서 계산된 c_new (cell state)와 h_new () 를 다음 시퀀스로 넘겨준다.
마지막으로 83~89번 줄에서 텐서플로 LSTM과 numpy LSTM의 forward pass 계산 결과를 비교하고 있다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
import tensorflow as tf
import numpy as np
num_input = 3
num_cell = 4
num_hidden = 5
seq_len = 10
forget_bias_value = 1
# TF LSTM
x = tf.placeholder(tf.float64, [1, seq_len, num_input]) # batch size = 1
c = tf.placeholder(tf.float64, [1, num_cell]) # batch size = 1
h = tf.placeholder(tf.float64, [1, num_hidden]) # batch size = 1
init_state = tf.contrib.rnn.LSTMStateTuple(c, h)
lstm = tf.contrib.rnn.LSTMCell(num_cell,
forget_bias = forget_bias_value,
num_proj=num_hidden)
out, state = tf.nn.dynamic_rnn(lstm, x,
sequence_length=np.ones([1])*seq_len, # batch size = 1
initial_state=init_state)
sess = tf.Session()
sess.run(tf.global_variables_initializer())
out_tf, state_ = sess.run([out, state],
feed_dict = {x: np.array([np.ones([seq_len, num_input])]),
c: np.array([np.linspace(0, 1, num=num_cell)]),
h: np.array([np.linspace(0.3, 0.8, num=num_hidden)])})
weights_tf = []
for w_ in range(len(lstm.weights)):
weights_tf.append( sess.run(lstm.weights[w_]) )
print('TF-LSTM')
print('\tc={}\n\th={}'.format(state_.c, state_.h))
# numpy LSTM
def sigmoid_array(x):
return 1 / (1+np.exp(-x))
x_np = np.array( np.ones([seq_len, num_input]) )
c_np = np.array( [np.linspace(0, 1, num=num_cell)] )
h_np = np.array( [np.linspace(0.3, 0.8, num=num_hidden)] )
out_np = []
for seq in range(seq_len):
args = np.concatenate(([x_np[seq]],h_np), axis=1)
weights = weights_tf[0]
biases = weights_tf[1]
if len(weights_tf) > 2:
weight_proj = weights_tf[2]
bias_proj = np.zeros([num_hidden])
out_t = np.matmul(args, weights)
concat = out_t + biases
i, j, f, o = np.split(concat, 4, 1)
c_tild = np.tanh(j)
forget_bias = forget_bias_value
sigmoid_f = sigmoid_array( f + forget_bias )
c_new = sigmoid_f * c_np + sigmoid_array(i) * c_tild
h_new = sigmoid_array(o) * np.tanh(c_new)
if len(weights_tf) > 2:
h_new = np.matmul(h_new, weight_proj) + bias_proj
out_np.append( h_new )
h_np = h_new
c_np = c_new
print('NP-LSTM')
print('\tc={}\n\th={}'.format(c_new, h_new))
# check code
for chk in range(len(out_np)):
on = out_np[chk]
ot = out_tf[0][chk]
diff = np.sum( np.abs(on-ot) )
if diff > 1e-3:
print('error')
Backpropagation of LSTM
위 그림에서 화살표는 forward pass에서 벡터의 흐름이고, 노드는 특정 연산을 의미한다. concatenation 연산() 제외한 나머지 노드들은 element wise 연산이다. 화살표 중간의 weight 매트릭스들은 훈련시켜야할 파라미터들이고 linear transformation (매트릭스, 벡터 곱셈)을 한다. 화살표의 파란색 글씨는 backpropagation시 계산해야할 gradient들이다. 위 그림에서 주의해야할 점이 있다. 를 프로젝션해서 생성한 를 출력으로 사용하고 있는데 이것을 다음 시퀀스의 입력으로 사용하지 않고 있다. 대신 프로젝션 하기 전의 를 다음 셀의 입력으로 보낸다. 즉, 로 와 의 크기가 같다.
그럼 우선 다시 LSTM의 forward pass 연산을 정리해보자.
그리고 loss 레이어를 softmax cross entropy로 정의하자. 즉,
이고, 이때 는 번째 시퀀스의 레이블이고 one hot encoding으로 되어있다.
이제 그래디언트를 구하자. 위 그림에 있는 표기법을 따라서 정리하면 아래와 같다.
가장 좌측 변의 표기는 그림의 표기와 동일하게 맞춘것이고 이들은 모두 loss 을 미분하는 것을 의미한다. ( 기호에 대한 설명은 밑에 있음) 여기서, 과 은 앞 시퀀스()에서부터 전달되어진 그래디언트들이다. 최초 이 그래디언트 값들은 영(zero)이다. 이고, 들도 비슷하다.
그럼 이것으로부터 파라미터(weights, biases)의 그래디언트는 아래와 같이 계산한다.
Training LSTM network
그럼 이제 LSTM을 numpy로 직접 구현하고 training 시켜보는 것까지 해보자. 주어진 문자열의 바로 다음에 올 문자열을 예측하는 LSTM 네트워크를 훈련 시켜볼 것이다. 트레이닝의 입력으로 일정 길이의 문자열 시퀀스를 주고 바로 다음 문자열을 타겟으로 준다. (타켓은 입력보다 한 시퀀스 느린 문자열이다. 코드의 252번줄에서 타겟을 어떻게 만들었는지 확인할 수 있다.) 테스트 단계에서는 첫 문자열을 입력으로 주고, 출력된 LSTM의 결과를 다음 시퀀스의 입력으로 준다. (이곳과 이곳을 참고하였음)
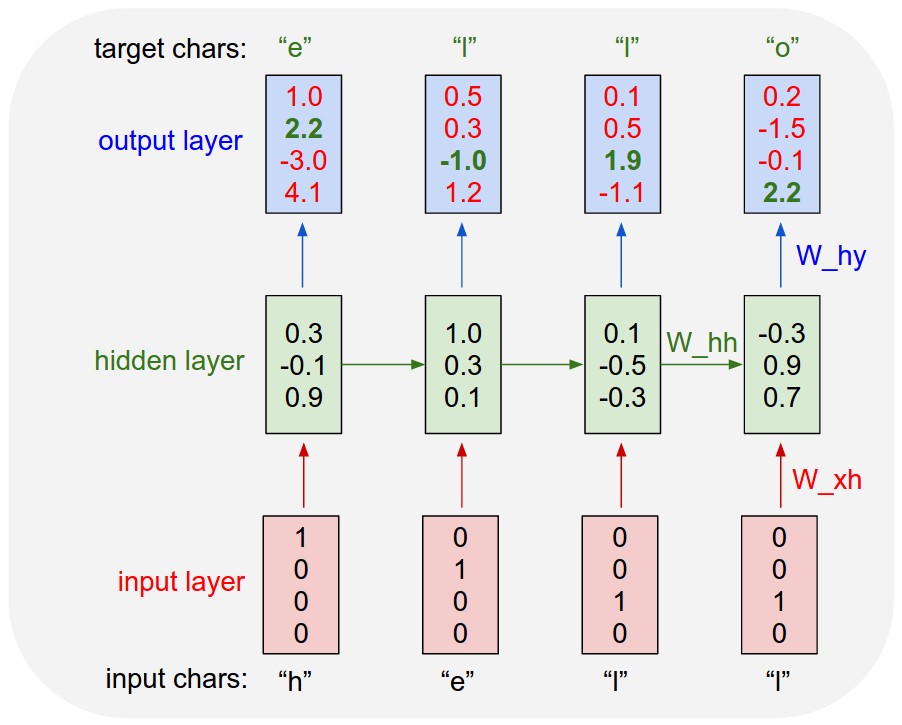
여기서 구현하고자 하는 것은 위 그림과 동일하며, 다만 가운데의 hidden layer가 RNN이 아닌 LSTM을 사용한다는 것만 다르다. 아래에서 사용한 input.txt 파일은 약 10만개의 문자열을 포함하고 있고 셰익스피어의 작품이다. (여기에서 다운 받을수 있음.)
여기서 numpy로 구현한 LSTM은 이다. 다만, 출력을 으로 맞추기 위한 레이어 ()가 있다.
backpropagation 관련 코드는 121~164 번줄에 있다.
211번 줄의 gradient_check 함수는 계산한(analytical) 그래디언트를 수치(numerical) 계산한 그래디언트와 비교하는 테스트 코드다.
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141
142
143
144
145
146
147
148
149
150
151
152
153
154
155
156
157
158
159
160
161
162
163
164
165
166
167
168
169
170
171
172
173
174
175
176
177
178
179
180
181
182
183
184
185
186
187
188
189
190
191
192
193
194
195
196
197
198
199
200
201
202
203
204
205
206
207
208
209
210
211
212
213
214
215
216
217
218
219
220
221
222
223
224
225
226
227
228
229
230
231
232
233
234
235
236
237
238
239
240
241
242
243
244
245
246
247
248
249
250
251
252
253
254
255
256
257
258
259
260
261
262
263
264
265
266
267
import numpy as np
from random import uniform
data = open('input.txt', 'r').read()
chars = np.sort(list(set(data)))
data_size, num_input = len(data), len(chars)
print("data has %d characters, %d unique" % (data_size, num_input))
char_to_idx = {ch: i for i, ch in enumerate(chars)}
idx_to_char = {i: ch for i, ch in enumerate(chars)}
# hyper-parameters
num_cell = 128
z_size = num_input + num_cell
seq_len = 29
learning_rate = 1e-4
forget_bias_value = 1
weight_init_sd = 0.1
# numpy LSTM
def sigmoid(x):
return(1 / (1 + np.exp( - x)))
def dsigmoid(y):
return y * (1 - y)
def tanh(x):
return np.tanh(x)
def dtanh(y):
return(1 - y * y)
parameters = {
'W_f': np.random.randn(num_cell, z_size) * weight_init_sd + 0.5,
'b_f': np.ones((num_cell, 1)) * forget_bias_value,
'W_i': np.random.randn(num_cell, z_size) * weight_init_sd + 0.5,
'b_i': np.zeros((num_cell, 1)),
'W_C': np.random.randn(num_cell, z_size) * weight_init_sd,
'b_C': np.zeros((num_cell, 1)),
'W_o': np.random.randn(num_cell, z_size) * weight_init_sd + 0.5,
'b_o': np.zeros((num_cell, 1)),
'W_v': np.random.randn(num_input, num_cell) * weight_init_sd,
'b_v': np.zeros((num_input, 1))
}
def calc_numerical_gradient(aparam, idx, delta, inputs, targets, h_prev, C_prev):
old_val = parameters[aparam].flat[idx]
x_t, C_t, h_t, y_t = {}, {}, {}, {}
h_t[-1] = np.copy(h_prev)
C_t[-1] = np.copy(C_prev)
# evaluate loss at [x + delta] and [x - delta]
parameters[aparam].flat[idx] = old_val + delta
loss_plus_delta = 0
for t in range(seq_len):
x_t[t] = np.zeros((num_input, 1))
x_t[t][inputs[t]] = 1
(_, _, _, _, C_t[t],
_, h_t[t], _, y_t[t]) = forward(x_t[t], h_t[t - 1], C_t[t - 1])
loss_plus_delta += - np.log(y_t[t][targets[t], 0])
x_t, C_t, h_t, y_t = {}, {}, {}, {}
h_t[-1] = np.copy(h_prev)
C_t[-1] = np.copy(C_prev)
parameters[aparam].flat[idx] = old_val - delta
loss_mins_delta = 0
for t in range(seq_len):
x_t[t] = np.zeros((num_input, 1))
x_t[t][inputs[t]] = 1
(_, _, _, _, C_t[t],
_, h_t[t], _, y_t[t]) = forward(x_t[t], h_t[t - 1], C_t[t - 1])
loss_mins_delta += - np.log(y_t[t][targets[t], 0])
parameters[aparam].flat[idx] = old_val #reset
grad_numerical = (loss_plus_delta - loss_mins_delta) / (2 * delta)
# Clip numerical error because analytical gradient is also clipped
[grad_numerical] = np.clip([grad_numerical], -1, 1)
return grad_numerical
def gradient_check(num_checks, delta, inputs, target, h_prev, C_prev, dParam):
# To calculate computed gradients
for aparam in parameters:
#Make a copy because this will get modified
d_copy = np.copy(dParam[aparam])
# Test num_checks times
for i in range(num_checks):
# Pick a random index
rnd_idx = int(uniform(0, d_copy.size))
grad_numerical = calc_numerical_gradient(aparam,
rnd_idx,
delta,
inputs,
target,
h_prev, C_prev)
grad_analytical = d_copy.flat[rnd_idx]
abs_err = abs(grad_analytical - grad_numerical)
# If relative error is greater than 1e-04
if abs_err > 1e-04:
print('Gradient error: %s (%f, %f) => %f'
%(aparam, grad_numerical, grad_analytical, abs_err))
def forward(x, h_prev, C_prev):
z = np.concatenate((h_prev, x), axis=0)
f = sigmoid(np.matmul(parameters['W_f'], z) + parameters['b_f'])
i = sigmoid(np.matmul(parameters['W_i'], z) + parameters['b_i'])
C_bar = tanh(np.matmul(parameters['W_C'], z) + parameters['b_C'])
C = f * C_prev + i * C_bar
o = sigmoid(np.matmul(parameters['W_o'], z) + parameters['b_o'])
h = o * tanh(C)
v = np.matmul(parameters['W_v'], h) + parameters['b_v']
y = np.exp(v) / np.sum(np.exp(v))
return z, f, i, C_bar, C, o, h, v, y
def backward(target, dh_latest, dC_latest, C_prev,
z, f, i, C_bar, C, o, h, v, y, dParam):
dv = np.copy(y)
dv[target] -= 1
dParam['W_v'] += np.matmul(dv, h.T)
dParam['b_v'] += dv
dh = np.matmul(parameters['W_v'].T, dv) + dh_latest
tanhC = tanh(C)
do = dh * tanhC
do = do * dsigmoid(o)
dParam['W_o'] += np.matmul(do, z.T)
dParam['b_o'] += do
dC = dh * o * dtanh(tanhC) + dC_latest
dC_bar = dC * i
dC_bar = dtanh(C_bar) * dC_bar
dParam['W_C'] += np.matmul(dC_bar, z.T)
dParam['b_C'] += dC_bar
di = dC * C_bar
di = di * dsigmoid(i)
dParam['W_i'] += np.matmul(di, z.T)
dParam['b_i'] += di
df = dC * C_prev
df = df * dsigmoid(f)
dParam['W_f'] += np.matmul(df, z.T)
dParam['b_f'] += df
dz = (np.matmul(parameters['W_f'].T, df) +
np.matmul(parameters['W_i'].T, di) +
np.matmul(parameters['W_C'].T, dC_bar) +
np.matmul(parameters['W_o'].T, do))
dh_pre = dz[:num_cell, :]
dC_pre = dC * f
return dh_pre, dC_pre
def train_one_sample(inputs, targets, h_start, C_start):
x_t, z_t, f_t, i_t = {}, {}, {}, {}
C_bar_t, C_t, o_t, h_t = {}, {}, {}, {}
v_t, y_t = {}, {}
h_t[-1] = np.copy(h_start)
C_t[-1] = np.copy(C_start)
loss = 0
for t in range(seq_len):
x_t[t] = np.zeros((num_input, 1))
x_t[t][inputs[t]] = 1
(z_t[t], f_t[t], i_t[t], C_bar_t[t], C_t[t],
o_t[t], h_t[t], v_t[t], y_t[t]) = forward(x_t[t], h_t[t - 1], C_t[t - 1])
loss += -np.log(y_t[t][targets[t], 0])
dParam = {
'W_f': np.zeros_like(parameters['W_f']),
'b_f': np.zeros_like(parameters['b_f']),
'W_i': np.zeros_like(parameters['W_i']),
'b_i': np.zeros_like(parameters['b_i']),
'W_C': np.zeros_like(parameters['W_C']),
'b_C': np.zeros_like(parameters['b_C']),
'W_o': np.zeros_like(parameters['W_o']),
'b_o': np.zeros_like(parameters['b_o']),
'W_v': np.zeros_like(parameters['W_v']),
'b_v': np.zeros_like(parameters['b_v'])}
dh_latest = np.zeros_like(h_start)
dC_latest = np.zeros_like(C_start)
for t in reversed(range(seq_len)):
dh_latest, dC_latest = backward(targets[t], dh_latest, dC_latest, C_t[t - 1],
z_t[t], f_t[t], i_t[t], C_bar_t[t], C_t[t], o_t[t], h_t[t], v_t[t], y_t[t], dParam)
# gradient clipping
for param in dParam:
np.clip(dParam[param], -1, 1, out=dParam[param])
# check gradient computation: analytical gradient vs numerical gradent
gradient_check(3, 1e-6, inputs, targets, h_start, C_start, dParam)
# parameter update, SGD
for param in dParam:
parameters[param] += - (learning_rate * dParam[param])
return loss, h_t[seq_len - 1], C_t[seq_len - 1]
def predict(h_start, C_start, initial_input, sentence_len):
x = np.zeros((num_input, 1))
x[initial_input] = 1
h_state = h_start
C_state = C_start
indexes = []
for t in range(sentence_len):
_, _, _, _, C_state, _, h_state, _, prob = forward(x, h_state, C_state)
#idx = np.argmax(prob.ravel())
idx = np.random.choice(range(num_input), p=prob.ravel())
x = np.zeros((num_input, 1))
x[idx] = 1
indexes.append(idx)
txt = ''.join(idx_to_char[ix] for ix in indexes)
return txt
if __name__ == '__main__':
data_ptr = 0
h_state = np.zeros((num_cell, 1))
C_state = np.zeros((num_cell, 1))
num_iter = 0
change_pt = 20000
smooth_loss = -np.log(1.0/num_input) * seq_len
while True:
if data_ptr + seq_len >= len(data):
data_ptr = 0
h_state = np.zeros((num_cell, 1))
C_state = np.zeros((num_cell, 1))
inputs = ([char_to_idx[ch] for ch in data[data_ptr:data_ptr + seq_len]])
targets = ([char_to_idx[ch] for ch in data[data_ptr + 1:data_ptr + seq_len + 1]])
loss, h_state, C_state = train_one_sample(inputs, targets, h_state, C_state)
smooth_loss = smooth_loss * 0.999 + loss * 0.001
# print(num_iter, loss)
if(num_iter%100) == 0 and num_iter > 0:
atext = predict(h_state, C_state, inputs[0], 200)
print('\n------------------------------------------------------')
print('{}th smooth_loss: {}, lr: {}'.format(num_iter, smooth_loss, learning_rate))
print('Sample Result:\n %s'%(atext,))
if(num_iter% change_pt) == 0 and learning_rate > 1e-6:
learning_rate*=0.9
data_ptr += np.random.randint(seq_len) + 1
num_iter += 1